Western blot


Il western blot o immunoblot è una tecnica biochimica che permette di identificare una determinata proteina in una miscela di proteine, mediante il riconoscimento da parte di anticorpi specifici;[1] in generale, per facilitare il riconoscimento, la miscela di proteine viene prima separata in base alle loro dimensioni (o peso molecolare) utilizzando un gel di poliacrilammide (ma esistono variazioni quali il dot blot o slot blot, in cui la miscela proteica non viene separata in base alle dimensioni, ma ci si affida alla selettività antigene/anticorpo); successivamente le proteine vengono trasferite su un supporto, che comunemente è una membrana di nitrocellulosa, e quindi si procede al riconoscimento vero e proprio della proteina mediante l'utilizzo di un anticorpo specifico.
Recentemente sono state messe a punto tecniche che permettono il riconoscimento antigene/anticorpo direttamente nella matrice del gel, evitando quindi il trasferimento su membrana. Questa tecnica si usa quando il campione è composto da una miscela di moltissime proteine tali che con una colorazione standard (blu di Coomassie o nitrato di argento) non si riuscirebbe a distinguerle l'una dall'altra, oppure quando, pur avendo bande ben distinte (discrete), la proteina di interesse è in quantità troppo basse per essere visualizzata con altre tecniche. Infatti il western possiede tre passaggi in cui avviene l'amplificazione del segnale, ciò rende visibili anche decimi di picomole (10−12mol) di proteina.[2][3][4]
Storia[modifica | modifica wikitesto]
Il metodo è stato ideato da George Stark alla Stanford University. Il nome Western blot fu dato dalla tecnica di W. Neal Burnette,[5] è un gioco sul nome Southern blot, tecnica per il rilevamento di DNA sviluppato precedentemente da Edwin Southern. Il rilevamento di RNA è chiamato Northern blot.[6] Ci sono molte aziende di reagenti specifici per anticorpi (monoclonali e policlonali) contro decine di migliaia di proteine differenti.[4] Gli anticorpi commerciali possono essere costosi, quelli non legati possono essere riutilizzati. Questo metodo è usato in biologia molecolare, biochimica, immunogenetica e altre discipline.
Fasi in un western blot[modifica | modifica wikitesto]
Preparazione tessuto[modifica | modifica wikitesto]
Campioni possono essere presi da tessuti interi o da colture cellulari. Tessuti solidi sono prima meccanicamente frantumati con frullatori (larghi volumi) o usando omogeneizzatori (volumi piccoli), o con sonicazione. Anche le cellule possono essere aperte con tali metodi. Virus o campioni prelevati da ambienti possono essere fonti di proteine e conseguentemente il western blot non è usato solo negli studi cellulari. Detergenti, sali, e soluzioni tampone possono essere impiegate per aumentare la lisi di cellule e solubilizzare proteine. Inibitori di proteasi e fosfatasi sono spesso aggiunte per prevenire la digestione dei campioni da parte dei loro stessi enzimi. La preparazione dei tessuti è spesso fatta a temperature basse per evitarne la degradazione. Una combinazione di tecniche biochimiche e meccaniche – comprese vari tipi di filtrazioni e centrifugazioni – possono essere usate per separare differenti cellule e organuli.
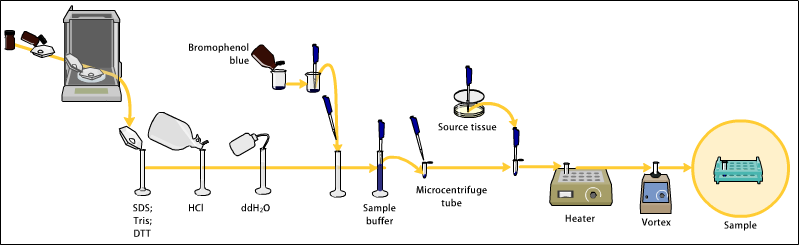
Elettroforesi su gel[modifica | modifica wikitesto]
Le proteine del campione sono separate usando l'elettroforesi in gel. La separazione di proteine può avvenire per punto isoelettrico (pI), peso molecolare, carica elettrica, o una combinazione di questi fattori. La natura della separazione dipende dal trattamento del campione e dalla natura del gel. Questo modo è il più usato per identificare una proteina. Il tipo più comune di elettroforesi in gel impiega gel di poliacrilammide (Elettroforesi su gel di poliacrilammide) e tamponi caricati con laurilsolfato di sodio (SDS). La SDS-PAGE (SDS polyacrylamide gel electrophoresis) mantiene polipeptidi in uno stato denaturato una volta trattato con agenti riducenti forti per rimuovere strutture secondarie e terziarie (esempio disolfuro [S-S] e gruppi mercaptani [SH e SH]) e permettere la separazione di proteine in base al loro peso molecolare. Le proteine campionate legano il SDS che conferisce loro carica negativa e si muovono in questo modo verso l'elettrodo positivo attraverso le rete di pori del gel. Le proteine più piccole migrano più velocemente attraverso questa rete e le proteine vengono separate in base alla dimensione (misurata in kiloDalton, kDa). La concentrazione di acrilammide determina la risoluzione del gel - maggiore la concentrazione, migliore la risoluzione di proteine a basso peso molecolare. Le proteine viaggiano solo in una dimensione lungo il gel per diversi blot. I campioni sono caricati in pozzetti nel gel. Una striscia è usualmente riservata per un marcatore o scala, una miscela commerciale di proteine con peso molecolare definito, tipicamente colorate così da formare bande colorate visibili. Quando la tensione elettrica è applicata lungo il gel, le proteine migrano attraverso esso a velocità differenti, in funzione della loro dimensione. Queste differenti velocità di avanzamento (mobilità elettroforetica) permettono la separazione in bande dentro ciascuna corsia.

È inoltre possibile usare l'elettroforesi bidimensionale.
Trasferimento[modifica | modifica wikitesto]
Per rendere accessibili le proteine alla rilevazione dell'anticorpo esse vengono trasferite dal gel ad una membrana di Nitrocellulosa o Polivinilidenfluoruro (PVDF). Il metodo principale per il trasferimento è chiamato "electroblotting" e usa corrente elettrica per trasferire le proteine dal gel alla membrana. Le proteine si muovono dal gel alla membrana mentre mantengono l'organizzazione presente nel gel. Un metodo più vecchio di trasferimento consiste nel mettere una membrana sul gel e strati di carta filtrante al di sopra di essa. Il tutto è posto in una soluzione tampone che si muove verso l'alto del filtro per capillarità, portando le proteine con sé. Quest'ultimo metodo non è usato perché richiede troppo tempo. L'electroblotting è preferito. Come risultato di entrambi i processi di "blotting", le proteine sono esposte su una superficie sottile per la rilevazione (vedi sotto). Membrane diverse sono scelte per le loro proprietà non-specifica di legame con proteine diverse. Il legame alla proteina è basato sulle interazioni idrofobiche, così come le interazioni tra la membrana e la proteina. Le membrane di nitrocellulosa sono più economiche di quelle in PVDF, ma molto più fragili e non facilmente riutilizzabili.
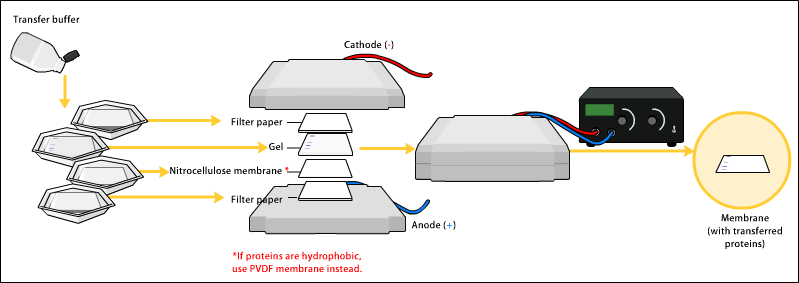
L'uniformità e l'effettivo trasferimento di proteina dal gel alla membrana può essere controllata colorando la membrana con Blu di Coomassie o con Ponceau S è il più comune dei due, data l'alta sensibilità e solubilità in acqua, più facilmente decolorabile per analizzare la membrana, come descritto sotto.[7]
Bloccaggio[modifica | modifica wikitesto]
Dopo aver scelto la membrana per la capacità di legare la proteina, è necessario prevenire le interazioni tra la membrana e l'anticorpo usato per rilevare la proteina bersaglio. Il bloccaggio di legami non specifici è raggiunto ponendo la membrana in una soluzione diluita di proteina - tipicamente 3-5% albumina sierica bovina (BSA) o latte in polvere in tampone Tris salino (TBS), con una piccola percentuale di detergente come Tween 20 o Triton X-100. La proteina nella soluzione diluita attecchisce alla membrana in tutti i punti dove le proteine bersaglio non hanno attecchito. Questo permette, quando l'anticorpo è aggiunto, che esso non si leghi a siti aspecifici in quanto già occupati dalla proteina precedentemente citata. Questo riduce il "rumore" nel prodotto finale, portando risultati più puliti, e eliminando falsi positivi.
Rilevamento[modifica | modifica wikitesto]
Durante il processo di rilevamento la membrana diventa il provino per la proteina d'interesse con un anticorpo modificato che è collegato a un enzima messaggero; quando esposto ad un appropriato substrato questo enzima crea una reazione colorimetrica e produce un colore. Per una serie di ragioni questo processo si svolge, tradizionalmente, in due fasi, anche se esistono metodi per processi a singola fase per determinate applicazioni.
Due fasi[modifica | modifica wikitesto]
- Anticorpo primario
Gli anticorpi primari sono generati quando la specie ospite o le colture di cellule immunitarie sono esposti alla proteina di interesse (o parte ad essa vicina). Normalmente questo è parte della risposta immunitaria, dove essi sono coltivati e usati come dispositivi specifici di rilevamento che legano la proteina direttamente. Dopo il bloccaggio, una soluzione diluita di anticorpo primario (generalmente tra 0.5 e 5 µg/ml) è incubato con la membrana agitandolo delicatamente. Tipicamente, la soluzione è composta da una soluzione tampone con una piccola percentuale di detergente, e qualche volta con latte in polvere o BSA. La soluzione anticorpo e la membrana possono essere sigillate e incubate assieme per 30 minuti o una notte intera. Può inoltre essere incubata a differenti temperature (a temperature più calde si associano legami più forti) sia sulla specifica proteina (target, il "segnale") e non-specifica ("rumore").
- Anticorpo secondario
Dopo il risciacquo della membrana per rimuovere l'anticorpo primario non legato, alla membrana è associato un altro anticorpo, diretto a una specifica specie di anticorpo primario. Anticorpi provengono da animali (o colture ibride di provenienza animale, ibridomi), un anticorpo secondario del topo si legherà a qualsiasi anticorpo primario di topo, permettendo risparmi sui costi, consentendo a un intero laboratorio di condividere un'unica fonte di anticorpo prodotto, e fornisce risultati migliori. Questo è conosciuto come anticorpo secondario, e data le sue proprietà bersaglio, ci si riferisce a "anti-topo," "anti-capra," ecc. L'anticorpo secondario è legato solitamente alla biotina o all'enzima messaggero come fosfatasi alcalina o perossidasi Armoracia rusticana. Questo significa che diversi anticorpi secondari si legheranno a un anticorpo primario, aumentando il segnale. Comunemente, un legame-perossidasi Armoracia rusticana secondario è usato per dividere un agente chemiluminescente, e la reazione prodotta emette luminescenza proporzionale all'ammontare di proteina. Una pellicola fotografica è posta contro la membrana; esposta alla luce della reazione crea un'immagine di anticorpi legato al blot. Un sistema più economico utilizza un colorante 4-cloronaftolo all'1% di perossido di idrogeno; la reazione di radicali perossido con il 4-cloronaftolo produce colore porpora scuro, permettendo la fotografia senza l'utilizzo di pellicole specifiche.

Come con l'ELISPOT e l'ELISA, l'enzima può essere dato da un substrato di molecole che può essere convertito dall'enzima in una prodotto di reazione colorata visibile sulla membrana (vedi le bande blu nella figura sotto). Un altro metodo con anticorpo secondario rilevatore utilizza un anticorpo fluoro-legato al vicino-infrarosso (NIR). La luce prodotta dall'eccitazione del colorante fluorescente è statica, permettendo un rilevamento accurato delle differenze di segnale prodotte da anticorpi etichetta legati a proteine sul western blot. Le proteine possono essere quantificate perché il segnale generato dalla differenza di quantità di proteine sulle membrane è misurata in modo statico, comparato alla chemiluminescenza, ove la luce è misurata in stato dinamico.[8] Una terza alternativa è usare etichette radioattive piuttosto che un enzima accoppiato all'anticorpo secondario, così come etichettare una proteina anticorpo non-legante come la proteina A di Staphylococcus o Streptavidina con un isotopo radioattivo di Iodio. Dato che altri metodi sono più sicuri, veloci, economici, questo è ora il meno usato; il vantaggio di questo metodo è la sensibilità radiografica che permette quantificazioni di proteina molto accurate, quando associato a software specifici (esempio l'Optiquant).
Singola fase[modifica | modifica wikitesto]
Storicamente il processo di campionamento fu fatto in due passi data la relativa semplicità di produrre anticorpi primari e secondari in processi separati. Questo dà ai ricercatori molti vantaggi in termini di flessibilità, e aggiunge una amplificazione nel rilevamento. Dato l'avvento di analisi proteiche ad alta efficienza e limiti più bassi di rilevamento, c'è stato interesse a sviluppare sistemi a singola fase che permettono maggior velocità di esecuzione e meno consumabili. Questo richiede un provino anticorpo che rileva la proteina d'interesse e che contenga un'etichetta rilevabile, provini che spesso sono disponibili per proteina bersaglio. Il provino primario è incubato con la membrana in un modo simile a quello dell'anticorpo primario in un processo a due fasi, e così è pronto per il rilevamento diretto dopo una serie di lavaggi.
Analisi[modifica | modifica wikitesto]
Dopo che i provini non legati sono lavati via, il western blot è pronto per la rilevazione di provini che sono etichettati e legati alla proteina di interesse. Non tutti i western blot rivelano proteine solo in una banda della membrana. Dimensioni approssimate sono prese dalla comparazione delle bande colorate rispetto al marcatore o scala caricata durante l'elettroforesi. Il processo è ripetuto per una proteina strutturale, come actina o tubulina, che non cambia tra i campioni. L'ammontare di proteina bersaglio è normalizzata alla proteina strutturale da controllare nei gruppi. Questa pratica assicura correzioni per l'ammontare della proteina totale sulla membrana, in caso di errori o trasferimenti incompleti.
Rilevazione colorimetrica[modifica | modifica wikitesto]
La rilevazione colorimetrica dipende dall'incubazione del western blot con un substrato che reagisce con l'enzima convertitore (come perossidasi) legato all'anticorpo secondario. Questo converte il colorante solubile in forma insolubile di differenti che precipitano in prossimità dell'enzima e conseguentemente colora la membrana. Lo sviluppo del blot è fermato dal lavaggio dei coloranti. I livelli di proteina sono valutati con densitometria (maggiore ove più colorato) o spettrofotometria.
Rilevazione con chemiluminescenza[modifica | modifica wikitesto]
I metodi con rilevazione chemiluminescente dipendono dall'incubazione del western blot con un substrato che emette luce quando esposto al convertitore dell'anticorpo secondario. La luce è rilevata da pellicola fotografica, e più recentemente da sensori CCD che catturano un'immagine digitale del western blot. L'immagine analizzata con densitometria rileva l'ammontare di proteina colorante e quantifica i risultati in termini di densità ottica. Nuovi software permettono ulteriori analisi come il peso molecolare.

Rilevazione radioattiva[modifica | modifica wikitesto]
I marcatori radioattivi non richiedono substrati enzimatici, permettendo il posizionamento di pellicole a raggi X direttamente contro il western blot che sviluppa, con l'esposizione, zone scure corrispondenti alla proteina d'interesse. È una tecnica dispendiosa e pericolosa, meglio preferire l'alternativa ECL (enhanced chemiluminescence).

Rilevazione fluorescente[modifica | modifica wikitesto]
Il provino fluorescente è eccitato dalla luce e l'emissione dell'eccitazione è rilevata da fotosensori come CCD equipaggiati di opportuni filtri che catturano un'immagine digitale del western blot; ciò permette ulteriori analisi come peso molecolare e un'analisi quantitativa del western blot. La rilevazione fluorescente è considerata uno dei migliori metodi per quantificare, anche se meno sensibile della chemiluminescenza.[9]
Prova secondaria[modifica | modifica wikitesto]
La maggior differenza tra membrane di nitrocellulosa e PVDF è la capacità di supportare l'eliminazione di anticorpi per il riutilizzo della membrana con provini successivi. Mentre ci sono protocolli ben definiti per membrane in nitrocellulosa, quelle in PVDF permettono riutilizzi numerosi prima di perdere sensibilità. Un'altra differenza è che, diversamente dalla nitrocellulosa, PVDF deve essere tenuto in soluzione al 95% di etanolo, isopropanolo o metanolo, prima dell'uso. Membrane PVDF tendono ad essere più sottili e robuste nell'uso.
Elettroforesi 2D[modifica | modifica wikitesto]
L'elettroforesi bidimensionale o elettroforesi 2D è un tipo di tecnica elettroforetica utilizzata nel campo della proteomica per separare miscele proteiche complesse (formate cioè da più specie proteiche differenti), come ad esempio una miscela di proteine estratta da una cellula in un dato momento del ciclo cellulare. La tecnica, come anticipa il nome, consta di due dimensioni ortogonali lungo le quali sono separate le diverse proteine. In proteomica, il termine ortogonale indica in genere due dimensioni lungo le quali la separazione avviene sfruttando principi fisici differenti, che non sono influenzati l'uno dall'altro. Nel caso dell'elettroforesi bidimensionale, i principi più utilizzati sono il punto isoelettrico e il peso molecolare.
Applicazioni medico diagnostiche[modifica | modifica wikitesto]
- L'HIV test impiega un western blot per rilevare anticorpi anti-HIV in un campione di plasma umano. Proteine da cellule da tessuto infettato da HIV sono separate e blotted su una membrana. Successivamente il siero da testare è applicato nella fase incubazione anticorpo primario; anticorpi liberi sono lavati via, si aggiunge un anticorpo secondario anti-umano collegato ad un segnale enzimatico. Le bande macchiate indicano le proteine nelle quali il siero del paziente contiene anticorpi.
- Un western blot è usato come test definitivo per l'encefalopatia spongiforme bovina (BSE, "mucca pazza").
- Alcune forme di malattia di Lyme vengono diagnosticate con western blot.
- Western blot può essere usato per confermare casi di epatite C.
- In medicina veterinaria, western blot è usato per confermare la positività da FIV nei gatti.
Protocolli[modifica | modifica wikitesto]
- Western Blotting Protocol from Protocolmonkey, su protocolmonkey.com.
- Modified western blotting protocols from Biotechniques, su e-biotek.com. URL consultato il 22 novembre 2012 (archiviato dall'url originale il 7 dicembre 2009).
- Dr. Mark Barton Frank Lab protocol, su protocol-online.org.
- Kamps's Western Blotting Protocol, su carmen.salk.edu. URL consultato il 22 novembre 2012 (archiviato dall'url originale l'11 dicembre 2012).
- Western blot analysis of subce, su natureprotocols.com. URL consultato il 22 novembre 2012 (archiviato dall'url originale il 20 aprile 2010).
- Western Blot Protocol, su prosci-inc.com. URL consultato il 22 novembre 2012 (archiviato dall'url originale il 14 novembre 2007).
- CSH Protocols collection: Immunoblotting, su cshprotocols.cshlp.org.
- western blot protocols, troubleshooting, scientific resources and research articles, su westernblotting.org. URL consultato il 22 novembre 2012 (archiviato dall'url originale l'11 settembre 2017).
- Western Blot Protocol from HowtoWesternBlot.net, su howtowesternblot.net.
Note[modifica | modifica wikitesto]
- ^ Ping-Chang Yang e Tahrin Mahmood, Western blot: Technique, theory, and trouble shooting, in North American Journal of Medical Sciences, vol. 4, n. 9, 2012, pp. 429–434, DOI:10.4103/1947-2714.100998, ISSN 1947-2714, PMC 3456489, PMID 23050259.
- ^ Towbin H, Staehelin T, Gordon J., Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications, in Proceedings of the National Academy of Sciences USA, vol. 76, n. 9, 1979, pp. 4350–54, DOI:10.1073/pnas.76.9.4350, PMC 411572, PMID 388439.
- ^ Renart J, Reiser J, Stark GR., Transfer of proteins from gels to diazobenzyloxymethyl-paper and detection with antisera: a method for studying antibody specificity and antigen structure, in Proceedings of the National Academy of Sciences USA, vol. 76, n. 7, 1979, pp. 3116–20, DOI:10.1073/pnas.76.7.3116, PMC 383774, PMID 91164.
- ^ a b Western blot antibody, su exactantigen.com. URL consultato il 29 gennaio 2009 (archiviato dall'url originale il 17 dicembre 2009).
- ^ Burnette WN., 'Western blotting': electrophoretic transfer of proteins from sodium dodecyl sulfate—polyacrylamide gels to unmodified nitrocellulose and radiographic detection with antibody and radioiodinated protein A, in Analytical Biochemistry, vol. 112, n. 2, 1981, pp. 195–203, DOI:10.1016/0003-2697(81)90281-5, ISSN 0003-2697, PMID 6266278.
- ^ Alwine J, Kemp D, Stark G, Method for detection of specific RNAs in agarose gels by transfer to diazobenzyloxymethyl-paper and hybridization with DNA probes, in Proceedings of the National Academy of Sciences USA, vol. 74, n. 12, 1977, pp. 5350–5354, Bibcode:1977PNAS...74.5350A, DOI:10.1073/pnas.74.12.5350, PMC 431715, PMID 414220.
- ^ Corley RB., A Guide to Methods in the Biomedical Sciences, Springer, 2005, pp. 11, ISBN 978-0-387-22844-0.
- ^ Ambroz K., Improving quantification accuracy for western blots (PDF), in Image Analysis, 20 settembre 2006 (archiviato dall'url originale il 29 agosto 2008).
- ^ Imaging Systems for Westerns: Chemiluminescence vs. Infrared Detection, 2009 - , Methods in Molecular Biology, Protein Blotting and Detection, vol. 536 (PDF) [collegamento interrotto], su springerlink.com, Humana Press. URL consultato il 26 febbraio 2010.
Voci correlate[modifica | modifica wikitesto]
Altri progetti[modifica | modifica wikitesto]
 Wikibooks contiene testi o manuali su la tecnica del western Blot
Wikibooks contiene testi o manuali su la tecnica del western Blot Wikimedia Commons contiene immagini o altri file su Western blot
Wikimedia Commons contiene immagini o altri file su Western blot
Collegamenti esterni[modifica | modifica wikitesto]
- Western blot analysis of sub-cellular fractionated samples using the Odyssey Infrared Imaging System (a protocol), su natureprotocols.com. URL consultato il 22 novembre 2012 (archiviato dall'url originale il 20 aprile 2010).
- Western blot protocol including example buffers and reprobing procedure, su prosci-inc.com. URL consultato il 22 novembre 2012 (archiviato dall'url originale il 14 novembre 2007).


