Condensina


Las condensinas son grandes complejos de proteínas que juegan importante papel en el ensamblado y segregación de los cromosomas durante la mitosis y meiosis.[1][2]
Composición de las subunidades[editar]
Condensinas en eucariotas[editar]

En las células de los vertebrados hay al menos dos tipos diferentes de complejos de condensinas, conocidos como condensina I y condensina II. Ambos complejos comparten el mismo par de subunidades centrales, SMC2 y SMC4, ambas pertenecientes a una numerosa familia de ATPasas cromosomales, conocidas como proteínas SMC. Las SMC (Proteínas de Mantenimiento Estructural por sus siglas en inglés) son proteínas encargadas del mantenimiento estructural de los cromosomas.[3] Cada uno de estos complejos contiene un conjunto distinto de subunidades reguladoras no-SMC.[4][5]
En los cultivos celulares de tejidos humanos, los dos complejos de condensinas son regulados de forma diferente durante el ciclo celular. La condensina II está presente dentro del núcleo celular durante la interfase y está involucrada en las etapas más tempranas de la condensación de cromosomas durante la profase del núcleo. Por otro lado, la condensina I está presente en el citoplasma durante la interfase, y gana acceso a los cromosomas solo luego de que la envoltura nuclear se rompe, al final de la profase. Durante la prometafase y la metafase, tanto la condensina I como la condensina II contribuyen a condensar y ensamblar los cromosomas, en donde las dos cromátidas hermanas son resueltas. Los dos complejos aparentemente permanecen asociados con los cromosomas luego que las cromátidas hermanas se separan, en la anafase. Al menos una de las subunidades de condensina I es uno de los blancos conocidos de las kinasas dependientes de ciclinas (Cdk).
| Complejo | Subunidad | Clasificación | S. cerevisiae | S. pombe | C. elegans | D. melanogaster | Vertebrados (Seres Humanos) |
|---|---|---|---|---|---|---|---|
| condensina I & II | SMC2 | ATPasa | Smc2 | Cut14 | MIX-1 | DmSmc2 | CAP-E (SMC2) |
| condensina I & II | SMC4 | ATPasa | Smc4 | Cut3 | SMC-4 | DmSmc4 | CAP-C (SMC4) |
| condensina I | CAP-D2 | HEAT | Ycs4 | Cnd1 | DPY-28 | CG1911 | CAP-D2 (NCAPD2) |
| condensina I | CAP-G | HEAT | Ycg1 | Cnd3 | CAP-G1 | cap-g | CAP-G (NCAPG) |
| condensina I | CAP-H | kleisina | Brn1 | Cnd2 | DPY-26 | barren | CAP-H (NCAPH) |
| condensina II | CAP-D3 | HEAT | - | - | HCP-6 | CG31989 | CAP-D3 (NCAPD3) |
| condensina II | CAP-G2 | HEAT | - | - | CAP-G2 | -? | CAP-G2 (NCAPG2) |
| condensina II | CAP-H2 | kleisina | - | - | KLE-2 | CG14685 | CAP-H2 (NCAPH2) |
| condensina IDC | variante SMC4 | ATPasa | - | - | DPY-27 | - | - |
La estructura y la función de la condensina I se ha conservado desde las levaduras a los humanos, pero las levaduras no tienen condensina II. En los nemátodos, la condensina II juega un rol importante en el ensamblado de los cromosomas y la segregación, donde el complejo de las proteínas relacionadas con la condensina I participan en la regulación génica de los cromosomas., i.e. compensación de dosis. Aún en bacterias, formas ancestrales de las condensinas regulan la organización y segregación de sus cromosomas (nucleoides).
Un complejo de proteínas distinto pero estructuralmente relacionado con la cohesina, está involucrado en la cohesión de cromátides hermanas. Contienen un par diferente de subunidades SMCs, SMC1 y SMC3, como sus subunidades ATPasa centrales.

Condensinas en procariotas[editar]
Algunas especies de procariotas también poseen complejos proteicos similares a las condensinas que juegan un papel importante en la organización y la segregación cromosómica. Los condensinas procariotas se pueden clasificar en dos tipos: SMC-ScpAB[6] y MukBEF.[7] Muchas especies de eubacterias y arqueas tienen SMC-ScpAB, mientras que un grupo de eubacterias (Gamma Proteobacterias) tienen MukBEF.
| Complejo | Subunidad | Clasificación | B. subtilis | Caulobacter | E.coli |
|---|---|---|---|---|---|
| SMC-ScpAB | SMC | ATPasa | SMC/BsSMC | SMC | - |
| SMC-ScpAB | ScpA | Kleisina | ScpA | ScpA | - |
| SMC-ScpAB | ScpB | ? | ScpB | ScpB | - |
| MukBEF | MukB | ATPasa | - | - | MukB |
| MukBEF | MukE | ? | - | - | MukE |
| MukBEF | MukF | Kleisina | - | - | MukF |
Mecanismos Moleculares[editar]
Actividades moleculares[editar]
La condensina I purificada presenta una tensión positiva en el ADN de cadena en espiral dependiente de la hidrólisis de ATP,[8] y también cuenta con una actividad de ATPasa estimulada por ADN in vitro. El dímero SMC2-SMC4 tiene la capacidad para alinear nuevamente (rehibridar) al ADN de una sola hebra complementaria, y esta actividad no requiere de hidrólisis de ATP.[9]
Estructuras Moleculares[editar]
Los dímeros de SMC que actúan como las subunidades de condensina centrales muestran una forma de V característica.[10][11]
Funciones mitóticas[editar]
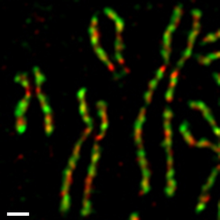
En las células cultivadas a partir de tejidos humanos, ambos complejos de condensinas son regulados diferencialmente durante el ciclo celular.[12][13] La condensina II se encuentra presente en el núcleo durante la interfase y participa en la condensación en etapa temprana de los cromosomas en el núcleo durante la profase. Por otra parte, la condensina I está presente en el citoplasma durante la interfase, y sólo puede acceder a los cromosomas después de romper la envoltura nuclear al final de la profase. Durante prometafase y metafase , tanto la condensina 1 como la 2 contribuyen al segundo conjunto de cromosomas condensados, que consta de dos cromátides hermanas. Los dos complejos permanecen asociados con los cromosomas al parecer después de que las cromátidas hermanas se separan una de otra durante la anafase. Se sabe que al menos una de las subunidades de la condensina I es un objetivo directo de una quinasa dependiente de ciclina (CDK).[14]
Modificaciones pos-traduccionales[editar]
Las subunidades de la condensina pueden sufrir, de manera dependiente del ciclo celular, una variedad de modificaciones post-traduccionales, entre ellas, la fosforilación ha sido la más estudiada. La Cdk1 (quinasa dependiente de ciclina 1) es una de las quinasas implicadas en la activación de la condensina, como así también hay una quinasa implicada en la regulación negativa como la CK2 (caseína quinasa 2).
| Complejo | Subunidad | Organismo | Sitio de fosforilación | Quinasa |
|---|---|---|---|---|
| Condensinas I y II | SMC4 | S. pombe | T19 | Cdk1 |
| S. cerevisiae | Varios | Cdk1 | ||
| Condensina Me | CAP-D2 | X. laevis | T1314, T1348, T1353 | Cdk1 |
| CAP-H | Humano | S570 | Cdk2 | |
| Humano | S70 | Aurora B | ||
| S. pombe | S5, S41, S52 | Aurora B | ||
| CAP-D2, -G, -H | Humano | - | Aurora B | |
| S. cerevisiae | Varios | Cd5 | ||
| Condensina II | CAP-D3 | Humano | T1415 | Cdk1 |
| Humano | S1419 | Pdk1 | ||
| CAP-G2 | Humano | TD1010 | - | |
| CAP-H2 | Humano | S492 | mps1 | |
| D. melanogaster | - | CK1a |
Referencias[editar]
- ↑ Hirano, Tatsuya (25 de febrero de 2016). «Condensin-Based Chromosome Organization from Bacteria to Vertebrates». Cell 164 (5): 847-857. ISSN 1097-4172. PMID 26919425. doi:10.1016/j.cell.2016.01.033. Consultado el 6 de diciembre de 2023.
- ↑ Hirano, Tatsuya (25 de febrero de 2016). «Condensin-Based Chromosome Organization from Bacteria to Vertebrates». Cell 164 (5): 847-857. ISSN 1097-4172. PMID 26919425. doi:10.1016/j.cell.2016.01.033. Consultado el 6 de diciembre de 2023.
- ↑ Schleiffer, Alexander; Kaitna, Susanne; Maurer-Stroh, Sebastian; Glotzer, Michael; Nasmyth, Kim; Eisenhaber, Frank (Marzo de 2003). «Kleisins: a superfamily of bacterial and eukaryotic SMC protein partners». Molecular Cell 11 (3): 571-575. ISSN 1097-2765. PMID 12667442. doi:10.1016/s1097-2765(03)00108-4. Consultado el 6 de diciembre de 2023.
- ↑ Hirano, T.; Kobayashi, R.; Hirano, M. (16 de mayo de 1997). «Condensins, chromosome condensation protein complexes containing XCAP-C, XCAP-E and a Xenopus homolog of the Drosophila Barren protein». Cell 89 (4): 511-521. ISSN 0092-8674. PMID 9160743. doi:10.1016/s0092-8674(00)80233-0. Consultado el 6 de diciembre de 2023.
- ↑ Ono, Takao; Losada, Ana; Hirano, Michiko; Myers, Michael P.; Neuwald, Andrew F.; Hirano, Tatsuya (3 de octubre de 2003). «Differential contributions of condensin I and condensin II to mitotic chromosome architecture in vertebrate cells». Cell 115 (1): 109-121. ISSN 0092-8674. PMID 14532007. doi:10.1016/s0092-8674(03)00724-4. Consultado el 6 de diciembre de 2023.
- ↑ Mascarenhas, Judita; Soppa, Jörg; Strunnikov, Alexander V.; Graumann, Peter L. (17 de junio de 2002). «Cell cycle-dependent localization of two novel prokaryotic chromosome segregation and condensation proteins in Bacillus subtilis that interact with SMC protein». The EMBO journal 21 (12): 3108-3118. ISSN 0261-4189. PMID 12065423. doi:10.1093/emboj/cdf314. Consultado el 6 de diciembre de 2023.
- ↑ Yamazoe, M.; Onogi, T.; Sunako, Y.; Niki, H.; Yamanaka, K.; Ichimura, T.; Hiraga, S. (1 de noviembre de 1999). «Complex formation of MukB, MukE and MukF proteins involved in chromosome partitioning in Escherichia coli». The EMBO journal 18 (21): 5873-5884. ISSN 0261-4189. PMC 1171653. PMID 10545099. doi:10.1093/emboj/18.21.5873. Consultado el 6 de diciembre de 2023.
- ↑ Kimura, K.; Hirano, T. (22 de agosto de 1997). «ATP-dependent positive supercoiling of DNA by 13S condensin: a biochemical implication for chromosome condensation». Cell 90 (4): 625-634. ISSN 0092-8674. PMID 9288743. doi:10.1016/s0092-8674(00)80524-3. Consultado el 6 de diciembre de 2023.
- ↑ Sutani, T.; Yanagida, M. (21 de agosto de 1997). «DNA renaturation activity of the SMC complex implicated in chromosome condensation». Nature 388 (6644): 798-801. ISSN 0028-0836. PMID 9285594. doi:10.1038/42062. Consultado el 6 de diciembre de 2023.
- ↑ Melby, T. E.; Ciampaglio, C. N.; Briscoe, G.; Erickson, H. P. (21 de septiembre de 1998). «The symmetrical structure of structural maintenance of chromosomes (SMC) and MukB proteins: long, antiparallel coiled coils, folded at a flexible hinge». The Journal of Cell Biology 142 (6): 1595-1604. ISSN 0021-9525. PMC 2141774. PMID 9744887. doi:10.1083/jcb.142.6.1595. Consultado el 6 de diciembre de 2023.
- ↑ Anderson, David E.; Losada, Ana; Erickson, Harold P.; Hirano, Tatsuya (4 de febrero de 2002). «Condensin and cohesin display different arm conformations with characteristic hinge angles». The Journal of Cell Biology 156 (3): 419-424. ISSN 0021-9525. PMC 2173330. PMID 11815634. doi:10.1083/jcb.200111002. Consultado el 6 de diciembre de 2023.
- ↑ Ono, Takao; Fang, Yuda; Spector, David L.; Hirano, Tatsuya (Julio de 2004). «Spatial and temporal regulation of Condensins I and II in mitotic chromosome assembly in human cells». Molecular Biology of the Cell 15 (7): 3296-3308. ISSN 1059-1524. PMID 15146063. doi:10.1091/mbc.e04-03-0242. Consultado el 6 de diciembre de 2023.
- ↑ Hirota, Toru; Gerlich, Daniel; Koch, Birgit; Ellenberg, Jan; Peters, Jan-Michael (15 de diciembre de 2004). «Distinct functions of condensin I and II in mitotic chromosome assembly». Journal of Cell Science 117 (Pt 26): 6435-6445. ISSN 0021-9533. PMID 15572404. doi:10.1242/jcs.01604. Consultado el 6 de diciembre de 2023.
- ↑ Kimura, K.; Hirano, M.; Kobayashi, R.; Hirano, T. (16 de octubre de 1998). «Phosphorylation and activation of 13S condensin by Cdc2 in vitro». Science (New York, N.Y.) 282 (5388): 487-490. ISSN 0036-8075. PMID 9774278. doi:10.1126/science.282.5388.487. Consultado el 6 de diciembre de 2023.
Véase también[editar]
Enlaces externos[editar]
 Wikimedia Commons alberga una categoría multimedia sobre Condensina.
Wikimedia Commons alberga una categoría multimedia sobre Condensina.- MeSH: condensin (en inglés)