Codón de terminación

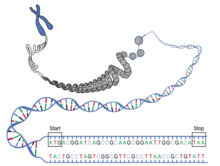
En genética se denomina codón de terminación, codón de parada, codón sin sentido o codón stop a aquel codón que no determina ningún aminoácido según el código genético. Su función es acotar el mensaje cifrado por el ADN que dará lugar al ARN mensajero; de este modo, limita en el extremo 3' el marco abierto de lectura de los genes.[1]
Su descubrimiento se debe a las observaciones de Sydney Brenner en el fago T4. Brenner estudiaba la expresión de una proteína de la cápside del virus, y encontró dos mutantes de ella, que producían proteínas más cortas que el silvestre y, que, además, en presencia de una mutación supresora en la célula de la bacteria hospedadora, recuperaban la longitud original. Analizando el aminoácido que habría de seguir tras el punto truncado de la proteína de la cápside, Brenner observó que se trataba de aquellos cuya mutación puntual en alguna de las pares de bases del triplete podía dar lugar a un codón «UAG» (adenina-uracilo-guanina) en el ARN mensajero. De este modo, intuyó que este codón no codificaba un aminoácido, sino una pausa (un punto o una coma) en el mensaje genético.[2]
Existen tres codones de terminación, que reciben distintos nombres. «UAG», el primero descubierto, se conoce como «codón ámbar»; «UGA», como «codón ópalo»; y «UAA», como «codón ocre».[3][4]
Su mecanismo de funcionamiento es el siguiente: cuando el ribosoma llega a uno de los codones de paro, entra al sitio A ribosomal un factor de terminación (RF1 o RF2 en bacterias, eRF en eucariotas) que impide la entrada de un nuevo ARNt, por lo que al aminoácido anterior ya no se le puede unir ninguno más y se libera, junto con todo el resto del péptido del que forma parte.
Los codones de paro, sin embargo, constituyen una excepción a la universalidad del código genético. En algunos genes de algunas especies no se interpretan como codones de paro, sino como nuevos aminoácidos que se suman a los 20 conocidos. Así, el codón UGA puede codificar la selenocisteína (cisteína con selenio en vez de azufre), y el codón UAG puede codificar la pirrolisina.
Véase también[editar]
Referencias[editar]
- ↑ Watson, J, D.; Baker, T. A.; Bell, S. P.; Gann, A.; Levine, M. et Losick, R (2004). Molecular Biology of the Gene (Fifth edition edición). San Francisco: Benjamin Cummings. ISBN 0-321-22368-3.
- ↑ Griffiths, J .F. A. et al. (2002). Genética. McGraw-Hill Interamericana. ISBN 84-486-0368-0.
- ↑ S. Brenner, A.O.W. Stretton, and S. Kaplan (1965). «Genetic Code: The 'Nonsense Triplets' for Chain Termination and their Suppression». Nature 206: 994-998. doi:10.1038/206994a0.
- ↑ S. Brenner, L. Barnett, E.R. Katz and F.H.C. Crick (1967). «UGA: A Third Nonsense Triplet in the Genetic Code». Nature 213: 449-450. doi:10.1038/213449a0.