تعلم الآلة في المعلوماتية الحية

تعلم الآلة (بالإنجليزية: Machine learning)، هو مجال فرعي من علم الحاسب الذي يشمل على تطوير خوارزميات تعلم كيفية إصدار التوقعات استنادا إلى البيانات، يحتوي على عدد من التطبيقات الناشئة في مجال المعلوماتية الحيوية. المعلوماتية الحيوية تتعامل مع طرق حسابية ورياضية النهج من أجل فهم ومعالجة البيانات البيولوجية.[1]
قبل ظهور خوارزميات تعلم الآلة، كان لابد من برمجة خوارزميات المعلوماتية الحيوية بشكل صريح باليد، والذي ثبت لمشاكل مثل تنبؤ بالبنية البروتينية انه صعب للغاية.[2] تقنيات تعلم الآلة مثل التعلم العميق يمكن الخوارزمية من استخدام تعلم الميزة التلقائي مما يعني أنه استنادا إلى البيانات وحدها، الخوارزمية يمكن أن تتعلم كيفية الجمع بين عدة ميزات من البيانات المدخلة إلى مجموعة من الميزات أكثر تجريدا والتي يمكن من خلالها إجراء مزيد من التعلم. تسمح طريقة تعلم الأنماط المتعددة الطبقات في البيانات المدخلة لهذه النظم لإصدار تنبؤات معقدة جداُ عندما تدرب على مجموعات كبيرة من البيانات. في السنوات الأخيرة، ارتفع حجم وعدد مجموعات البيانات البيولوجية المتاحة، مما مكن الباحثين في مجال المعلوماتية الحيوية من الاستفادة من أنظمة تعلم الآلة هذه. تم تطبيق تعلم الآلة على ستة مجالات فرعية رئيسية للمعلوماتية الحيوية: الجينوم، بروتيوميات، نسق مايكروي، نظم علم الأحياء، تطور، والتنقيب في النصوص.[3]
التطبيقات[عدل]
علم الجينوم[عدل]
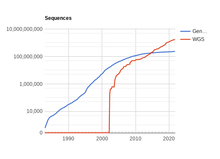
ينطوي علم الجينوم على دراسة الجينوم، التسلسل الكامل للحمض النووي، للكائنات الحية. في حين أن بيانات التسلسل الجينومي كانت متفرقة تاريخياً بسبب الصعوبة التقنية في تسلسل قطعة من الحمض النووي، فإن عدد التسلسلات المتاحة يتزايد بشكل تصاعدي.[4] ومع ذلك، في حين أن البيانات الخام أصبحت متاحة وسهلة المنال بشكل متزايد، إلا أن التفسيرالبيولوجي لهذه البيانات يحدث بوتيرة أبطأ بكثير.[5] لذلك، هنالك حاجة متزايدة لتطوير أنظمة تعلم الآلة التي يمكنها بشكل تلقائي تحديد موقع جينات ترميزالبروتين في تسلسل حمض نووي معين. هذه مشكلة في علم الأحياء الحسابي المعروف باسم التنبؤ الجيني.
عادة ما يتم التنفيذ الآلي الجيني من خلال مزيج من ما يعرف باسم عمليات البحث الخارجية والداخلية.[5] بالنسبة إلى البحث الخارجي، يتم تمرير تسلسل الحمض النووي المدخل عبر قاعدة بيانات كبيرة من التسلسلات التي تم اكتشافها جيناتها سابقًا وتعليم موقعها. يمكن تحديد عدد من جينات التسلسل من خلال تحديد أي سلاسل من القواعد ضمن التسلسل تتطابق مع تسلسلات جينية معروفة. ولكن نظراً لحدود حجم قاعدة البيانات للتسلسلات الجينية المعروفة، لا يمكن تحديد جميع الجينات في تسلسل معين من خلال التشابه فقط. لذلك، هناك حاجة إلى بحث داخلي حيث يحاول برنامج التنبؤ الجيني تحديد الجينات المتبقية من تسلسل الحمض النووي وحده.
كما تم استخدام تعلم الآلة لمشكلة تراصف السلسلة المتعدد والتي تنطوي على محاذاة العديد من تسلسل الحمض النووي أو الأحماض الأمينية من أجل تحديد مناطق التشابه التي يمكن أن تشير إلى تاريخ تطوري مشترك. ويمكن أيضًا استخدامه لاكتشاف وتصور إعادة ترتيب الجينوم.[6]
بروتيوميات[عدل]

البروتينات، سلاسل الأحماض الأمينية، تكتسب الكثير من وظائفها من تطوي البروتين، حيث تتطابق إلى بنية ثلاثية الأبعاد. تتكون هذه البنية من عدد من طبقات الطي، بما في ذلك البنية الأساسية (أي السلسلة المسطحة من الأحماض الأمينية)، والبنية الثانوية (صفائح ألفا وأوراق بيتا)، والبنية الثالثة، والبنية الرابعة.
المراجع[عدل]
- ^ Chicco D (ديسمبر 2017). "Ten quick tips for machine learning in computational biology". BioData Mining. ج. 10 ع. 35: 1–17. DOI:10.1186/s13040-017-0155-3. PMC:5721660. PMID:29234465.
{{استشهاد بدورية محكمة}}: صيانة الاستشهاد: دوي مجاني غير معلم (link) - ^ Yang، Yuedong؛ Gao، Jianzhao؛ Wang، Jihua؛ Heffernan، Rhys؛ Hanson، Jack؛ Paliwal، Kuldip؛ Zhou، Yaoqi. "Sixty-five years of the long march in protein secondary structure prediction: the final stretch?". Briefings in Bioinformatics. DOI:10.1093/bib/bbw129. مؤرشف من الأصل في 2017-09-18.
- ^ Larrañaga، Pedro؛ Calvo، Borja؛ Santana، Roberto؛ Bielza، Concha؛ Galdiano، Josu؛ Inza، Iñaki؛ Lozano، José A.؛ Armañanzas، Rubén؛ Santafé، Guzmán. "Machine learning in bioinformatics". Briefings in Bioinformatics: 86–112. DOI:10.1093/bib/bbk007. مؤرشف من الأصل في 2017-09-18.
- ^ "GenBank and WGS Statistics". www.ncbi.nlm.nih.gov (بالإنجليزية). Archived from the original on 2019-04-28. Retrieved 2017-05-06.
- ^ أ ب Mathé، Catherine؛ Sagot، Marie-France؛ Schiex، Thomas؛ Rouzé، Pierre (1 أكتوبر 2002). "Current methods of gene prediction, their strengths and weaknesses". Nucleic Acids Research. ج. 30 ع. 19: 4103–4117. DOI:10.1093/nar/gkf543. ISSN:1362-4962. PMC:140543. PMID:12364589.
- ^ Pratas، D؛ Silva, R؛ Pinho, A؛ Ferreira, P (18 مايو 2015). "An alignment-free method to find and visualise rearrangements between pairs of DNA sequences". Scientific Reports (Group Nature). ج. 5 ع. 10203: 10203. Bibcode:2015NatSR...510203P. DOI:10.1038/srep10203. PMC:4434998. PMID:25984837.