إطار القراءة

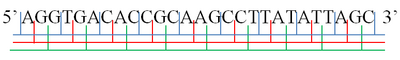
A·GGT·GAC·ACC·GCA·AGC·CTT·ATA·TTA·GC
AG·GTG·ACA·CCG·CAA·GCC·TTA·TAT·TAG·C
في البيولوجيا الجزيئية، إطار القراءة هو طريقة لتقسيم وقراءة تسلسل النوكليوتيدات في جزيء حمض نووي (دنا أو رنا) إلى مجموعات متتالية وغير متداخلة من النوكليوتيدات الثلاثية التي تسمى كودونات .[1] يتكون كل كودون (رامزة) من ثلاث نوكليوتيدات ويرمز إلى حمض أميني معين أو إشارة توقف أثناء الترجمة.
لدى كل سلسلة واحدة من جزيء الحمض النووي نهاية فسفوريل تسمى النهاية 5'، ونهاية هيدروكسيل تسمى النهاية 3' وتحدد هاتين النهايتين الاتجاه 3'→5' الذي يتم النسخ وفقه. توجد ثلاثة إطارات قراءة يمكن للريبوسوم أن يبدأ القراءة منها، ويبدأ كل إطار قراءة من إحدى النوكليوتيدات الثلاث في الكودون. في حالة الحمض النووي مزدوج السلاسل، توجد ثلاث إطارات قراءة إضافية أخرى يمكن قراءتها من السلسلة المكملة في الاتجاه 3'→5' الخاص بها. جزيء الحمض النووي مزدوج السلاسل متوازي ومتضاد في الإتجاه، وبالتالي فإن الإتجاه 3'→ 5' وفق السلسلة الثانية يتوافق مع الإتجاه 5'→ 3' وفق السلسلة الأولى.[2][3]
بشكل عام وفي معظم الحالات، إطارُ قراءةٍ واحدٍ فقط من الإطارات الثلاثة الموجودة في جزء من سلسلة حمض نووي هو من يكون ذو صلة بيولوجية (إطار قراءة مفتوح)، أي يحمل شيفرة يمكن أن ينتج عنها ناتج جيني. ومع ذلك، توجد بعض النسخ الفيروسية التي يمكن ترجمتها باستخدام عدة إطارات قراءة متداخلة.[4] يوجد مثال معروف على إطاري قراءة متداخلين في دنا متقدرة الثدييات: حيث يتداخل جزءا جينين مشفرين لوحدتين فرعين من إنزيم الأتِباز.
الشيفرة الجينية[عدل]

يُشفر الدنا تسلسل الأحماض الأمينية في البروتينات عبر سلسلة من الكودونات ثلاثية النوكليوتيدات. يمكن قراءة أي تسلسل من الدنا بست طرق مختلفة: ثلاث إطارات قراءة في السلسلة الأولى (يبدأ كل إطار من نوكليوتيد مختلف) وثلاث إطارت قراءة في السلسلة الثانية بالاتجاه المعاكس. أثناء النسخ يقرأ بوليميراز الرنا سلسلة الدنا القالب في الاتجاه 5′→3′ لكن الرنا الرسول يُخلَّق في الاتجاه 3′→5′.[5] الرنا الرسول هو سلسلة مفردة وبالتالي لا يحتوي سوى على ثلاث إطارات قراءة ممكنة، والتي لا يُترجم منها سوى إطار واحد. تُترجم كودونات الرنا الرسول وفق الاتجاه 3′→5′ إلى أحماض أمينية بواسطة الريبوسوم لإنتاج سلسلة عديد ببتيد.
إطار القراءة المفتوح[عدل]
إطار القراءة المفتوح ويرمز له بـ (أورف، ORF) هو إطار قراءة أو جزء منه لديه احتمالية أن يُنسَخ إلى رنا ويترجم إلى بروتين. ويتكون من كودون بدء يليه تسلسلٌ مستمر من النوكليوتيدات التي يكون طولها عادة قابلا للقسمة على ثلاثة وينتهي بكودون توقف في نفس إطار القراءة.
عندما ينتج تسلسل أحماض أمينية من ترجمة افتراضية لإطار قراءة مفتوح ويبقى غير معروف (أي غير محدد على مستوى البروتين تجريبيا ولم يتم استخلاصه ومعرفة وظيفته)، فإن إطار القراءة المفتوح الخاص بهذا التسلسل كان يسمى إطار قراءة غير معروف (URF) وهو مصطلح قديم كان العلماء يستخدمونه عند تحديد تسلسلات جينومات المتقدرات والبلاستيدات الخضراء وتم التخلي عنه لصالح مصطلح إطار القراءة المفتوح الذي ينطبق على جميع أقسام الدنا. حسب التعريف: إطار القراءة المفتوح غير مُعرّف ويمكن أن يكون جينا محتملا [الإنجليزية] ويمكن أن لا يكون، وحين يتم تعريفه فذلك يعني أن ناتجه معروف وأنه أصبح بذلك جينا حقيقيا.[6] على سبيل المثال: سُمي الجين MT-ATP8 أول مرة بـ URF A6L حين تم تحديد التسلسل الكامل لجينوم المتقدرة.[7]

إطارات قراءة متعددة[عدل]
يؤدي استخدام إطارات قراءة متعددة إلى إمكانية وجود جينات متداخلة، توجد العديد من هذه الجينات في الفيروسات، بدائيات النوى، وجينومات المتقدرات.[8] تستخدم بعض الفيروسات مثل فيروس التهاب الكبد ب وفيروس اصفرار وتقزم الشعير عدة جينات متداخلة في إطارات قراءة مختلفة.
في حالات نادرة، يمكن أن ينزاح الريبوسوم من إطار قراءة إلى آخر أثناء ترجمة رنا رسول (انزياح الإطار الريبوسومي) ويتسبب ذلك في ترجمة الجزء الأول من الرنا الرسول وفق إطار قراءة، والجزء الثاني وفق إطار قراءة آخر مختلف. الانزياح الريبوسومي مختلف عن طفرة انزياح الإطار -التي يتغير فيها إطار القراءة بسبب حذف أو غرز- لأن تسلسل نوكليوتيدات الرنا أو الدنا عند الانزياح الريبوسومي لا يتغير وإنما إطار القراءة فقط.
مراجع[عدل]
- ^ "معلومات عن إطار القراءة على موقع jstor.org". jstor.org. مؤرشف من الأصل في 2020-04-17.
- ^ Rainey S، Repka J. "Quantitative sequence and open reading frame analysis based on codon bias" (PDF). Systemics, Cybernetics and Informatics. ج. 4 ع. 1: 65–72. مؤرشف من الأصل (PDF) في 2020-08-06.
- ^ Badger JH، Olsen GJ (أبريل 1999). "CRITICA: Coding Region Identification Tool Invoking Comparative Analysis". Mol Biol Evol. ج. 16 ع. 4: 512–24. DOI:10.1093/oxfordjournals.molbev.a026133. PMID:10331277. مؤرشف من الأصل في 2019-12-20.
- ^ Lander، Eric. "MITx 7.00x Biology". مؤرشف من الأصل في 2019-12-07.
- ^ Lodish (2007). Molecular Cell Biology (ط. 6th). W. H. Freeman. ص. 121. ISBN:978-1429203142.
- ^ "Unidentified Reading Frame according to wiley online library". wiley online library. مؤرشف من الأصل في 2020-10-31. اطلع عليه بتاريخ 2020-10-27.
- ^ Anderson S، Bankier AT، Barrell BG، de Bruijn MH، Coulson AR، Drouin J، Eperon IC، Nierlich DP، Roe BA، Sanger F، Schreier PH، Smith AJ، Staden R، Young IG (أبريل 1981). "Sequence and organization of the human mitochondrial genome". Nature. ج. 290 ع. 5806: 457–65. Bibcode:1981Natur.290..457A. DOI:10.1038/290457a0. PMID:7219534.
- ^ Johnson Z، Chisholm S (2004). "Properties of overlapping genes are conserved across microbial genomes". Genome Res. ج. 14 ع. 11: 2268–72. DOI:10.1101/gr.2433104. PMC:525685. PMID:15520290.
| في كومنز صور وملفات عن: إطار القراءة |